1. RandomForest 기본 예시 코드
from sklearn.ensemble import RandomForestRegressor
# 랜덤 포레스트 모델 초기화
rf_model = RandomForestRegressor()
rf_model.fit(X_train, y_train)
pred = rf_model.predict(X_test)
2. RandomForest 파라미터 삽입 예시 코드
from sklearn.ensemble import RandomForestRegressor
# 랜덤 포레스트 모델 초기화
rf_model = RandomForestRegressor(n_estimators=100, max_features="sqrt", random_state=42)
rf_model.fit(X_train, y_train)
pred = rf_model.predict(X_test)
3. RandomForest 하이퍼파라미터 튜닝 예시 코드
from sklearn.model_selection import GridSearchCV
params = {
'n_estimators':[2800, 3000, 3300],
'max_depth' : [6, 8, 10, 12],
'min_samples_leaf' : [8, 12, 18],
'min_samples_split' : [8, 16, 20]
}
rf_clf = RandomForestRegressor(random_state=0, n_jobs=-1)
grid_cv = GridSearchCV(rf_clf , param_grid=params , cv=2, n_jobs=2, verbose=2 )
grid_cv.fit(x_train , y_train) # grid.cv.fit(train_x, train_y)
estimator =grid_cv.best_estimator_
pred = estimator.predict(x_valid) # estimator.predict(test)
print('최적 하이퍼 파라미터:\n', grid_cv.best_params_)
print('최고 예측 정확도: {0:.4f}'.format(grid_cv.best_score_))
evaluation(y_valid , pred)
4. RandomForest 하이퍼 파라미터
class sklearn.ensemble.RandomForestRegressor(
n_estimators=100,
max_features=1.0,
criterion='squared_error',
max_depth=None,
min_samples_split=2,
min_samples_leaf=1,
min_weight_fraction_leaf=0.0,
max_leaf_nodes=None,
min_impurity_decrease=0.0,
bootstrap=True,
n_jobs=None,
random_state=None,
verbose=0
)class sklearn.ensemble.RandomForestClassifier(
n_estimators=100,
max_features='sqrt',
criterion='gini',
max_depth=None,
min_samples_split=2,
min_samples_leaf=1,
min_weight_fraction_leaf=0.0,
max_leaf_nodes=None,
min_impurity_decrease=0.0,
bootstrap=True,
n_jobs=None,
random_state=None,
verbose=0,
class_weight=None
)
4.1. n_estimators: 생성할 트리의 개수 (int, default=100)
일반적으로 더 많은 트리를 사용할수록 모델의 성능이 향상됩니다.
다만 트리 개수가 일정 수준을 넘어서면 성능 향상의 폭이 급격히 줄어들면서 계산 비용만 증가하는 현상이 나타납니다.
실제 프로젝트에서는 성능과 효율성의 균형점을 찾는 것이 중요하며, 보통 100에서 1000 사이의 값을 실험해보는 것이 좋습니다.
4.2. max_features: 각 노드를 분할할 때 고려할 특성의 무작위로 선택되는 특성의 개수
회귀: {“sqrt”, “log2”, None}, int or float, default=1.0 (전체 특성 수)
분류: {“sqrt”, “log2”, None}, int or float, default="sqrt" (전체 특성 수의 제곱근)값을 작게 설정하면 분산 감소, 편향 증가, 복잡도 감소, 모델의 성능 감소
값을 크게 설정하면 분산 증가, 편향 감소, 복잡도 증가, 모델의 성능 증가, 과적합 위험
4.3. criterion: 노드의 분할의 품질을 평가하는 기준을 설정
회귀 문제에서는 "squared_error"가 기본값으로, 평균 제곱 오차를 기반으로 최적의 분할점을 찾습니다.
회귀 {“squared_error”, “absolute_error”, “friedman_mse”, “poisson”}, default=”squared_error”- "squared_error" (평균 제곱 오차, Mean Squared Error): 각 분할에서 평균 예측값과 실제값 간의 제곱 차이를 계산하고, 이를 모든 분할에 대해 합산하여 평균을 구합니다.
- "friedman_mse":(Friedman의 평균 제곱 오차): "squared_error"와 비슷한 개념이지만, 잠재적인 분할에 대한 Friedman의 개선 점수를 고려합니다. 이 개선 점수는 각 분할의 오차 감소 정도를 측정하여 최적의 분할을 선택하는 데 활용됩니다.
- "absolute_error" (평균 절대 오차, Mean Absolute Error): 각 분할에서 평균 예측값과 실제값 간의 절대 차이를 계산하고, 이를 모든 분할에 대해 평균을 구합니다. 이상치에 덜 민감합니다.
- "poisson" (포아송 손실, Poisson Loss): 각 분할에서 포아송 분포와 실제값 간의 편차를 측정합니다.
분류: {“gini”, “entropy”, “log_loss”}, default=”gini”- "gini": gini 지수가 낮을수록 노드의 순도가 높아짐
- "entropy": entropy가 낮을수록 노드의 순도가 높아짐
▶ 보통은 기본값인 "gini" (또는 "mse")가 잘 동작하지만, 특정 문제에 따라 다른 criterion 값을 실험하여 최적의 성능을 얻을 수도 있습니다.
4.4.max_depth: 트리의 최대 깊이 (int, default=None)
max_depth는 개별 트리의 최대 깊이를 제한하여 과적합을 방지하는 파라미터입니다.
기본값은 None으로, 이는 트리가 완전히 성장할 때까지 분할을 계속한다는 의미입니다.
깊이를 제한하면 모델이 단순해져서 일반화 성능이 향상될 수 있지만, 너무 얕게 설정하면 데이터의 복잡한 패턴을 포착하지 못할 수 있습니다. 일반적으로 5에서 20 사이의 값을 실험해보며, 교차 검증을 통해 최적값을 찾는 것이 좋습니다.
4.5.min_samples_split: 노드를 분할하기 위해 필요한 최소한의 샘플 개수 (int or float, default=2)
min_samples_split은 노드를 분할하기 위해 필요한 최소 샘플 수를 지정합니다.
기본값은 2이며, 이 값을 증가시키면 트리가 더 보수적으로 분할되어 과적합을 방지할 수 있습니다.
작은 값으로 설정하면 트리가 더 세밀하게 분할되어 복잡한 패턴을 학습할 수 있지만, 노이즈에 민감해질 수 있습니다.
데이터셋의 크기와 노이즈 수준을 고려하여 적절한 값을 선택해야 합니다.
4.6.min_samples_leaf: 노드를 분할하기 위해 리프 노드가 가져야 할 최소한의 샘플 개수(int or float, default=1)
min_samples_leaf는 리프 노드가 가져야 할 최소 샘플 수를 제한합니다.
기본값은 1이지만, 이를 증가시키면 모델이 더 일반화된 예측을 하게 됩니다.
특히 노이즈가 많은 데이터에서는 이 값을 2 이상으로 설정하는 것이 도움이 될 수 있습니다
4.7.max_leaf_nodes: 생성될 수 있는 최대 리프 노드의 개수를 제한 (int, default=None)
일반적으로 랜덤 포레스트의 트리는 자라다가 가능한 한 많은 리프 노드를 생성하려는 경향이 있습니다.
그러나 max_leaf_nodes를 설정함으로써 트리의 성장을 제한할 수 있습니다.
max_leaf_nodes를 지정하면 트리는 해당 개수 이상의 리프 노드를 생성하지 않습니다.
이를 통해 과적합을 방지하고, 일반화 능력을 향상시킬 수 있습니다.
작은 max_leaf_nodes 값은 모델의 단순성을 증가시키고 편향을 높일 수 있습니다.
4.8.min_impurity_decrease: 노드를 분할하기 위한 최소한의 불순도 감소량.(float, default=0.0)
노드의 분할은 해당 분할이 전체 트리의 불순도를 감소시키는지 여부에 따라 결정
min_impurity_decrease는 불순도 감소량이 이 값보다 크거나 같을 때 분할이 이루어져야 함을 의미.
즉, 분할이 불순도 감소량이 충분히 크지 않으면 노드는 더 이상 분할되지 않고 잎 노드로 유지.
값을 작게 설정하면 노드의 분할이 더 자주 일어남.(양수로 지정) 모델의 복잡도 증가
값이 크게 설정하면 노드의 분할이 덜 발생. 제약이 강해짐
4.9.n_jobs: 모델 학습 및 예측에 사용되는 병렬 작업의 수(int, default=None)
랜덤 포레스트는 결정 트리의 앙상블로 구성되어 있으며, 각각의 결정 트리는 독립적으로 학습 및 예측이 가능합니다.
이러한 특성을 활용하여 n_jobs 파라미터를 설정함으로써 병렬 처리를 통해 모델의 학습 및 예측 속도를 향상시킬 수 있습니다.
파라미터
- 1: 모든 가능한 프로세서를 사용하여 병렬 처리합니다.
- 양의 정수: 지정된 수의 프로세서를 사용하여 병렬 처리합니다.
4.10.bootstrap: 부트스트래핑은 중복을 허용하여 샘플을 랜덤 하게 선택하는 방식, 모델의 다양성을 증가. (bool, default=True)
부트스트랩 샘플링의 사용 여부를 결정합니다. 기본값은 True이며, 이는 각 트리가 원본 데이터에서 중복을 허용하여 무작위로 선택된 샘플로 학습된다는 의미입니다. 이러한 무작위성은 개별 트리 간의 다양성을 증가시켜 앙상블 효과를 극대화합니다.
4.11.random_state: 랜덤 시드를 지정하는 파라미터로, 재현성을 위해 사용됩니다. (int, default=None)
random_state는 모델의 재현성을 보장하기 위한 랜덤 시드를 설정합니다. 동일한 값을 설정하면 항상 같은 결과를 얻을 수 있어, 실험의 일관성을 유지하고 결과를 비교하는 데 도움이 됩니다. 연구나 프로덕션 환경에서는 반드시 설정하는 것이 좋습니다.
4.12.verbose: 학습과 예측 시에 출력되는 정보의 상세도를 제어
- verbose=0: 출력이 비활성화되어 가장 적은 양의 정보만 표시.
- verbose=1: 학습 및 예측 과정에서 진행 상황 메시지 표시. 진행 상황을 모니터링하고 싶을 때 유용.
5.RandomForest 속성
5.1.best_estimator_
GridSearchCV 객체에서 최적의 추정기(estimator)를 반환하는 속성입니다.
GridSearchCV는 주어진 하이퍼파라미터 그리드(param_grid)에서 모든 조합을 시도하고, 교차 검증을 통해 각 조합의 성능을 평가합니다. 그리드 서치가 완료되면 best_estimator_ 속성은 최적의 조합으로 학습된 추정기(estimator)를 반환합니다.
grid_cv.best_estimator_> RandomForestRegressor(n_estimators = 3000, max_depth=8, min_samples_leaf=8, min_sample_split=8, n_jobs=-1, random_state=0)
5.2.feature_importances_
랜덤 포레스트는 다수의 의사 결정 트리를 앙상블하여 모델을 구성합니다. 각 트리는 피처들을 사용하여 분할을 수행하고, 이 과정에서 피처들의 중요도를 계산합니다. feature_importances_ 속성은 이러한 중요도 값을 모아서 제공합니다.
주로, 중요도 값은 0과 1 사이의 실수 값으로 표현되며, 피처들 간의 상대적인 중요도를 나타냅니다. 중요도 값이 클수록 해당 피처가 예측에 더 큰 영향을 미친다고 해석할 수 있습니다.
import matplotlib.pyplot as plt
import seaborn as sns
%matplotlib inline
ftr_importances_values = estimator.feature_importances_
ftr_importances = pd.Series(ftr_importances_values,index=x_train.columns)
ftr_top = ftr_importances.sort_values(ascending=False)
plt.figure(figsize=(8,6))
plt.title('Feature importances')
sns.barplot(x=ftr_top , y = ftr_top.index)
plt.show()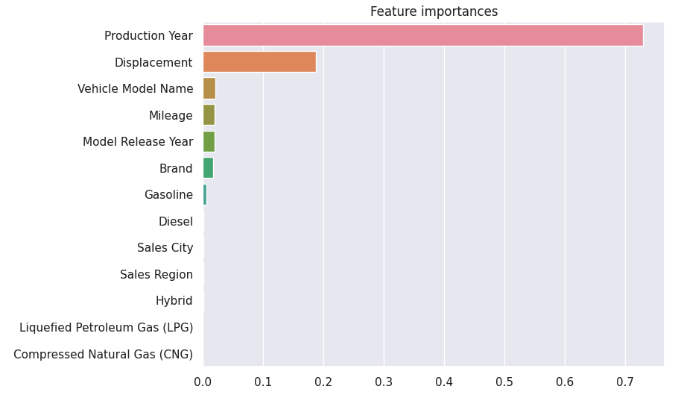
5.3. n_features_in_
RandomForest 모델이 학습 시에 관찰한 특성(feature)의 수를 나타내는 속성입니다.
이 속성은 모델이 fit() 메서드를 사용하여 학습된 후에 사용할 수 있습니다.
grid_cv.n_features_in_> 16
5.4.feature_names_in_
RandomForest 모델이 학습 시에 사용된 특성(feature)의 이름을 나타내는 속성입니다.
이 속성은 입력 데이터(X)의 특성 이름이 모두 문자열로 되어 있을 때만 해당 속성을 사용할 수 있습니다.
"feature_names_in_"은 각 특성의 이름을 포함하는 배열 또는 리스트로 표현됩니다.
grid_cv.feature_names_in_> array(['생산년도', '모델출시년도', '브랜드', '차량모델명', '판매도시', '판매구역', '주행거리', '배기량', '압축천연가스(CNG)', '경유', '가솔린', '하이브리드', '액화석유가스(LPG)'], dtype=object)
6.RandomForest 공식 문서
RandomForestClassifier
Gallery examples: Probability Calibration for 3-class classification Comparison of Calibration of Classifiers Classifier comparison Inductive Clustering OOB Errors for Random Forests Feature transf...
scikit-learn.org
'pythonML' 카테고리의 다른 글
| [pythonML] 앙상블(ensemble model)이란? | 앙상블의 종류 | 편향 (Bias)과 분산(Variance) (1) | 2024.08.27 |
|---|---|
| [pythonML] XGBoost 하이퍼파라미터 (0) | 2023.06.13 |
| [pythonML] classification Metrics (분류 평가지표) (1) | 2023.01.26 |
| [pythonML] 회귀 트리 (0) | 2022.07.16 |
| [pythonML] 회귀- 다항회귀 (0) | 2022.07.16 |